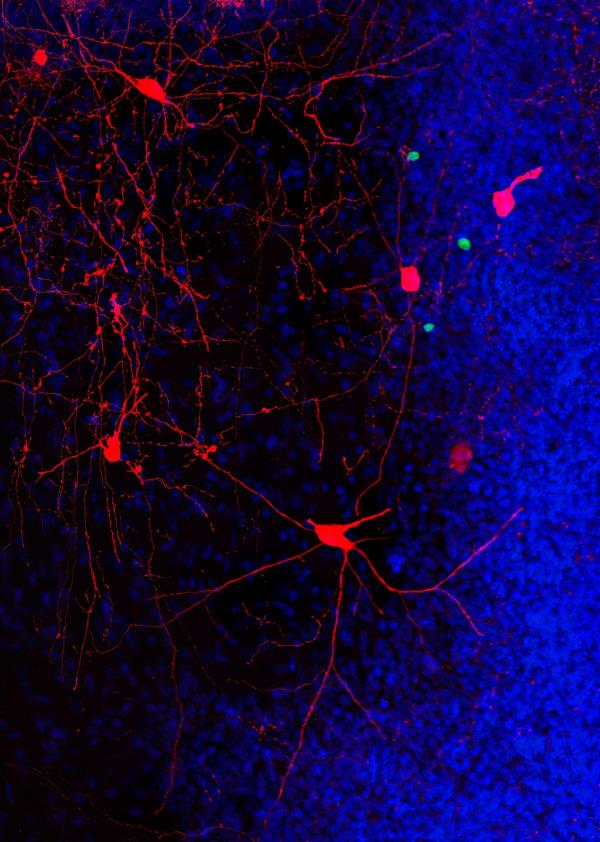
研究人员设计了一种计算分析方法来检测和区分体细胞结构变化的功能影响。
癌症是一种复杂多样的疾病,其相关突变的范围非常广泛。个体中这些基因组变化的组合被称为他们的“突变景观”。这些情况因癌症类型的不同而不同,即使是患有同一类型癌症的个体也可能有非常不同的突变模式。
科学家们已经记录了几种癌症的突变情况。体细胞结构变异(SVs)已被发现占所有致癌突变的50%以上。随着时间的推移,这些突变在细胞中发生,例如在细胞分裂过程中通过复制DNA中的错误,导致染色体结构的改变。
它们不是遗传的,只存在于受影响的细胞和它们的子细胞中。随着我们年龄的增长,这样的基因组改变变得越来越多,一个人的突变景观越来越像一个独特的马赛克。
尽管体细胞sv在癌症发展中起着至关重要的作用,但对它们的了解相对较少。“目前还缺乏分析它们对细胞功能影响的方法,”Max Delbrück中心基因组稳定性和体细胞镶嵌实验室负责人阿什利·桑德斯博士解释说。桑德斯最近与欧洲分子生物学实验室(EMBL)一起在《自然生物技术》杂志上发表了新的研究发现,这种情况正在改变。
她说:“我们开发了一种计算分析方法来检测和识别体细胞sv的功能影响。”这使研究小组能够了解不同白血病患者个体体细胞突变的分子后果,使他们对突变特异性改变有了新的认识。桑德斯说,也有可能利用这些发现来开发针对突变细胞的治疗方法,并补充说:“它们为个性化医疗开辟了令人兴奋的新途径。”
甚至比co更详细常规单细胞分析
他们的计算是基于来自Strand-seq的数据,这是一种特殊的单细胞测序方法,桑德斯在开发过程中发挥了重要作用,并于2012年首次引入科学界。这项技术可以比传统的单细胞测序技术更详细地检查细胞基因组。得益于复杂的实验方案,Strand-seq方法可以独立分析父母的两条DNA链(一条来自父亲,一条来自母亲)。
用传统的测序方法,区分这些同源物——形状和结构相似但不完全相同的染色体——几乎是不可能的。桑德斯解释说:“通过解析细胞内的个体同源物,可以比其他方法更好地识别体细胞sv。”研究人员和她的同事在2020年发表在《自然生物技术》杂志上的一篇论文中描述了这种方法。
该研究团队是柏林卫生研究所(Charité (BIH), Charité - Universitätsmedizin柏林和Max Delbrück中心)联合研究重点“个性化医疗的单细胞方法”的一部分。
在这项工作的基础上,他们现在还能够确定核小体在每个细胞中的位置。核小体是包裹在组蛋白蛋白复合物周围的DNA单元,在染色体组织中起着至关重要的作用。核小体的位置可以在基因表达过程中改变,包裹的类型揭示了基因是否活跃。桑德斯和她的同事开发了一种自我学习算法,以比较有和没有体细胞SV突变的患者细胞的基因活性,使他们能够确定结构变异的分子影响。
癌症治疗的新目标
桑德斯解释说:“我们现在可以从病人身上提取样本,寻找导致疾病的突变,还可以了解致病突变破坏的信号通路。”例如,该团队能够在一位白血病患者身上识别出一种罕见但非常具有侵略性的突变。核小体分析为研究人员提供了有关信号通路的信息,他们利用这些信息专门抑制含有突变的细胞的生长。桑德斯说:“这意味着一次测试就能告诉我们癌症形成过程中涉及的细胞机制。”“我们最终可以利用这些知识来开发个性化的治疗方法,以每个患者的独特情况为指导。”
引用:Hyobin Jeong, Karen Grimes, Kerstin K. Rauwolf, Peter-Martin Bruch, Tobias Rausch, Patrick Hasenfeld, Eva Benito, Tobias Roider, Radhakrishnan Sabarinathan, David Porubsky, Sophie A. Herbst, Büşra Erarslan-Uysal, john - christoph Jann, Tobias Marschall, Daniel Nowak, Jean-Pierre Bourquin, Andreas E. Kulozik, Sascha Dietrich, Beat Bornhauser, Ashley D. Sanders和Jan O. Korbel, 2022年11月24日,自然生物技术。DOI: 10.1038 / s41587 - 022 - 01551 - 4
由Ashley D. Sanders, Sascha Meiers, Maryam Ghareghani, David Porubsky, Hyobin Jeong, M. Alexandra C. C. van Vliet, Tobias Rausch, Paulina richter - pechazynska, Joachim B. Kunz, Silvia Jenni, Davide Bolognini, Gabriel M. C. Longo, Benjamin Raeder, Venla Kinanen, Jürgen Zimmermann, Vladimir Benes, Martin Schrappe, Balca R. Mardin, Andreas E. Kulozik, Beat Bornhauser, Jean-Pierre Bourquin,Tobias Marschall和Jan O. Korbel, 2019年12月23日,自然生物技术。DOI: 10.1038 / s41587 - 019 - 0366 - x